dcyang摄影小站

德钦 · 云南
梅里雪山日照金山

雨崩 · 云南
凌晨的神女峰

雨崩 · 云南
傍晚,雨崩冰湖返回途中

丽江·云南
路边摆摊的纳西族老奶奶

丽江·云南
黑龙潭公园远眺玉龙雪山

雨崩·云南
雨崩尼农线进村途中

雨崩·云南
路边休息,背后土坡,不知道啥时候来的骡子大哥

雨崩·云南
与自然融为一体的玛尼堆,很漂亮

建德·杭州
新安江畔嬉戏的小朋友

建德·杭州
天黑前,雨雾中的新安江

建德·杭州
新安江畔,雨中驻足

建德·杭州
雨中漫步新安江

哈尔滨·黑龙江
早市售卖大葱的阿姨们

哈尔滨·黑龙江
太阳岛公园的秋色

哈尔滨·黑龙江
松花江畔玩闹的人们

哈尔滨·黑龙江
早上的太阳岛,阳光甚好

吉林
泼水成冰

吉林
松花江边升起的太阳淹没在浓浓江雾中

吉林
冒着严寒来看雾凇的人们
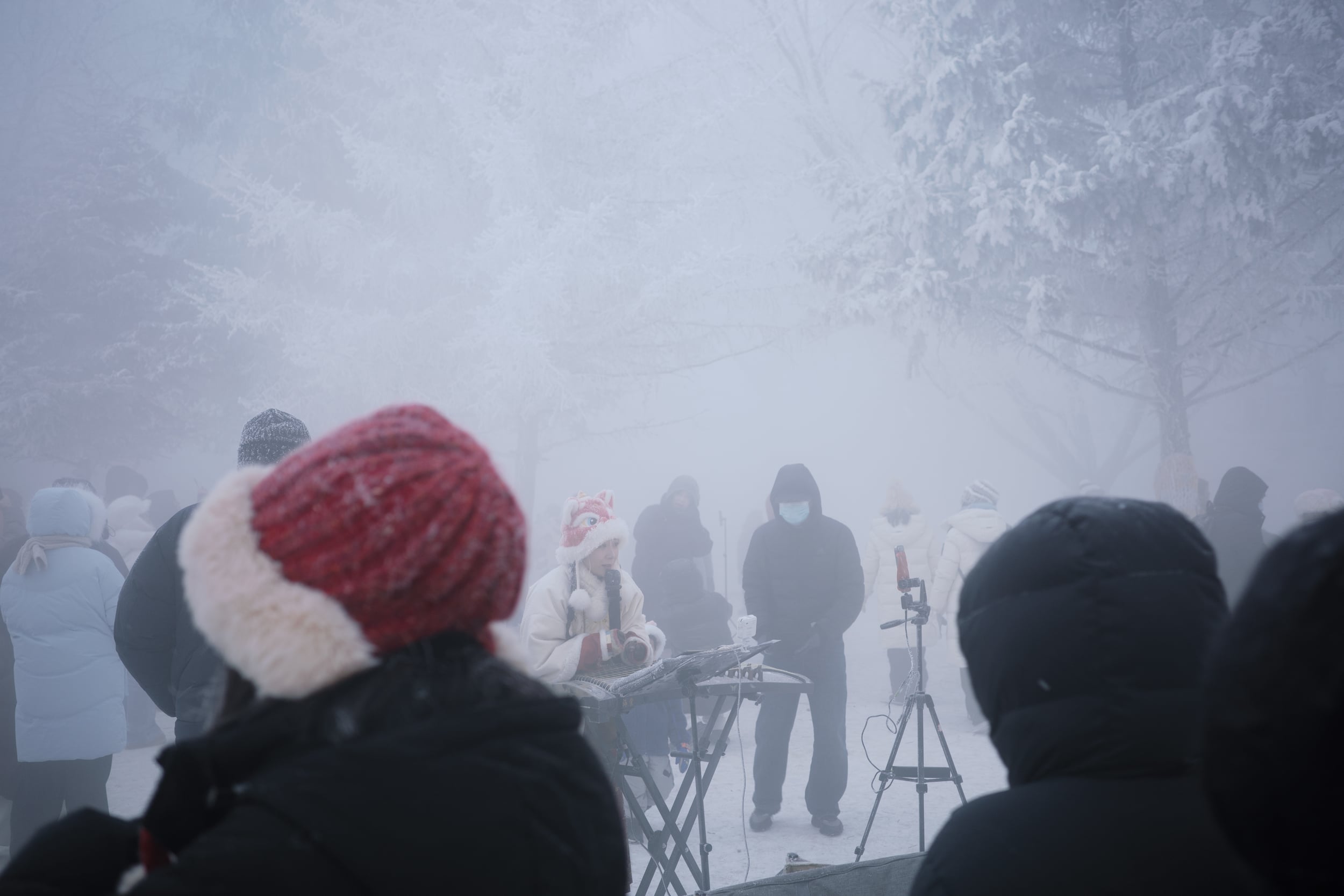
吉林
小雾凇岛上直播的小姐姐

吉林
真的是万缕银丝啊

吉林
路边的雾凇景观

昆明·云南
漂亮的小狗

昆明·云南
路边一角

昆明·云南
翠湖公园

昆明·云南
翠湖公园

青岛·山东
海军博物馆

青岛·山东
晒晒太阳,吹吹海风

青岛·山东
和谐的画面

青岛·山东
静静的车车

青岛·山东
崂山民宿边的风景,山海相连

青岛·山东
山海日出

丽水
露个脸

丽水
瀑布,小桥,还有人

北京
北海公园入口,仿佛穿越回清朝

北京
结冰的湖面

北京
北海公园一角

德钦 · 云南
梅里雪山日照金山